Images Numériques¶
Les images numériques sont des images décrites dans un format numérique. On peut les utiliser pour interpréter quantitativement certaines grandeurs. Cette partie dresse un rapide tableau des différentes approches basiques qui permettent d’effectuer ces tâches et donne des pistes pour aller plus loin sur chaque thème abordé.
Les points abordés ici sont détaillés plus finement dans le cours: Traitement_Signal_slides.pdf
Formation¶
Selon le dispositif qui l’a produite, le sens physique des informations contenues dans une image est différent. Voici quelques exemples d’images classées par type d’informations:
- Lumière visible: photographie , microscopie optique.
- Lumière infrarouge: thermographie.
- Electrons: microscopie électronique.
- Topologie: microscopie à force atomique.
- Et de nombreux autres…
Structure¶
Deux grandes familles d’images numériques existent:
- Images vectorielles : elles constituées de figures géométriques (droites, polygones, …). Elles sont idéales pour représenter des schémas et des courbes.
- Images matricielles : elles sont constituées d’une matrice de pixels. Chaque pixel d’une même image porte le même type d’informations. Ces informations sont scindées en canaux chacun contenant un nombre qui peut être entier (généralement 8 bits) ou des flottant dans le cas d’images scientifiques. Il est important de noter que la couleur d’un pixel tel qu’il apparait quand on représente une image n’est pas associé de manière unique à l’information contenue dans le pixel. Par exemple, dans une photographie, on cherche à ce que la représentation du pixel soit fidèle à la vision humaine et donc on va donc la décomposer en 3 canaux (rouge, vert, bleu par exemple) avec eventuellement un quatrième canal destiné à coder la transparence. On parle alors d’image polychrome. A l’opposé dans une image à vocation scientifique, on cherchera généralement à quantifier un phénomème scientifique par un seul canal, si possible sous forme flottante. On parle alors d’image monochrome.
On prend un exemple de photographie: grenouille.jpg
from PIL import Image
import numpy as np
import matplotlib.pyplot as plt
im = Image.open(
'../data/grenouille.jpg')
fig = plt.figure(0)
plt.clf()
plt.imshow(im, origin = "lower")
plt.xlabel("pixels")
plt.ylabel("pixels")
plt.show()
(Source code, png, hires.png, pdf)

On s’intéresse maintenant uniquement à des images monochromes formées de nombres flottants. Ainsi si on dispose d’une photographie, on peut isoler un canal ou construire une combinaison quelquconque de canaux comme suit.
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
im = Image.open('../data/grenouille.jpg')
rouge, vert, bleu = im.split()
rouge = np.array(rouge)
vert = np.array(vert)
bleu = np.array(bleu)
fig = plt.figure(0) # On cree une figure
plt.clf()
ax1 =fig.add_subplot(3,1,1)
plt.imshow(rouge, origin = "upper")
plt.xticks([])
plt.yticks([])
plt.grid()
cbar = plt.colorbar()
cbar.set_label("Pixel value")
plt.ylabel("Rouge")
plt.title("Canaux")
ax2 = fig.add_subplot(3,1,2)
plt.imshow(vert, origin = "upper")
plt.xticks([])
plt.yticks([])
plt.grid()
cbar = plt.colorbar()
cbar.set_label("Pixel value")
plt.ylabel("Vert")
ax3 = fig.add_subplot(3,1,3)
plt.imshow(bleu, origin = "upper")
plt.xticks([])
plt.yticks([])
plt.grid()
cbar = plt.colorbar()
cbar.set_label("Pixel value")
plt.ylabel("Bleu")
plt.show()
(Source code, png, hires.png, pdf)
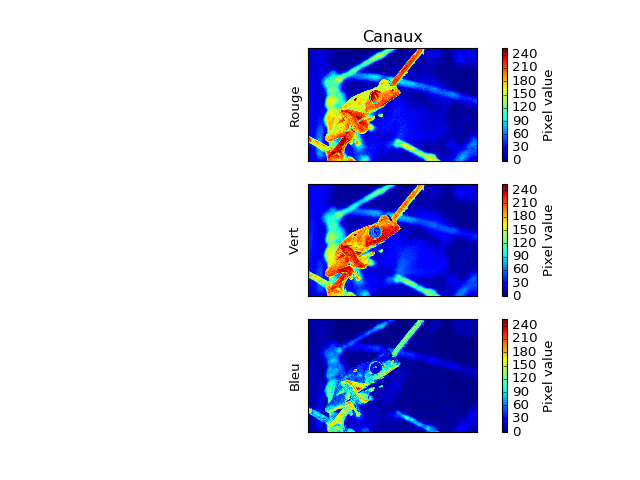
Une image se résumera donc à une matrice \(Z_{ij}\) où \(i\) et \(j\) sont les indices des pixels. Dans certains cas, on pourra ajouter des informations comme les coordonnées \(X_{ij}\) et \(Y_{ij}\) des pixels. Toutes ces matrices sont décrites dans le format Python numpy.array avec des pixels sous forme numpy.float64.
Operations¶
Dans cette partie, nous utiliserons aussi une image générée pour l’occasion:
(Source code, png, hires.png, pdf)
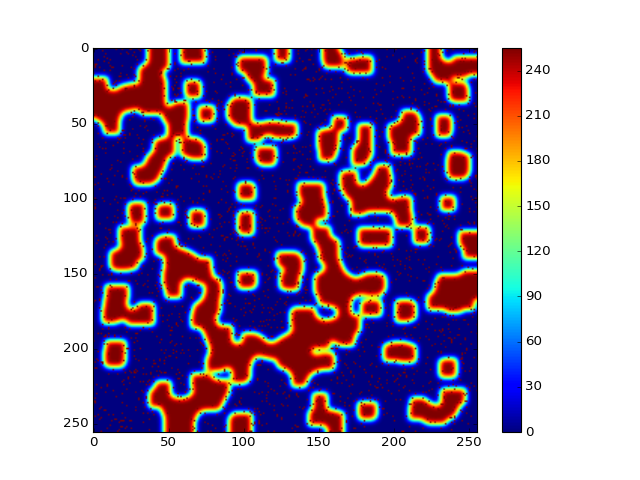
Vous pouvez télécharger l’image ici: image.jpg
Lecture¶
from PIL import Image
import numpy as np
import matplotlib.pyplot as plt
im = Image.open(
'../data/grenouille.jpg')
fig = plt.figure(0)
plt.clf()
plt.imshow(im, origin = "lower")
plt.xlabel("pixels")
plt.ylabel("pixels")
plt.show()
(Source code, png, hires.png, pdf)

Sauvegarde¶
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
from matplotlib import cm
im = Image.open('../data/grenouille.jpg')
rouge, vert, bleu = im.split()
z = np.array(rouge)
z = np.uint8(cm.copper(z)*255)
im2 = Image.fromarray(z)
im2.save("grenouille_saved.jpg")
Rognage¶
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
from matplotlib import cm
im = Image.open('../data/grenouille.jpg')
rouge, vert, bleu = im.split()
z = np.array(rouge)
ny, nx = z.shape
cx, cy = 200, 250
zc = z[cx:-cx, cy:-cy]
nyc, nxc = zc.shape
fig = plt.figure(0) # On cree une figure
plt.clf()
ax1 =fig.add_subplot(3,1,1)
plt.imshow(z, origin = "upper")
plt.xticks([0, nx])
plt.yticks([0, ny])
ax2 = fig.add_subplot(3,1,2)
plt.imshow(zc, origin = "upper", interpolation = "nearest")
plt.xticks([0, nxc])
plt.yticks([0, nyc])
plt.show()
(Source code, png, hires.png, pdf)
Rotations¶
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
from matplotlib import cm
from scipy import ndimage
im = Image.open('../data/grenouille.jpg')
rouge, vert, bleu = im.split()
z = np.array(rouge)
zrr = ndimage.rotate(z, 30.)
zrn = ndimage.rotate(z, 30.,
reshape = False)
ny, nx = z.shape
nyrr, nxrr = zrr.shape
nyrn, nxrn = zrn.shape
fig = plt.figure(0) # On cree une figure
plt.clf()
ax1 =fig.add_subplot(3,1,1)
plt.imshow(z, origin = "upper")
plt.xticks([0, nx])
plt.yticks([0, ny])
ax2 = fig.add_subplot(3,1,2)
plt.imshow(zrr, origin = "upper")
plt.xticks([0, nxrr])
plt.yticks([0, nyrr])
ax2 = fig.add_subplot(3,1,3)
plt.imshow(zrn, origin = "upper")
plt.xticks([0, nxrn])
plt.yticks([0, nyrn])
plt.show()
(Source code, png, hires.png, pdf)
Histogramme¶
Un histogramme représente la répartition de la population de pixels en fonction de leur altitude. Une valeur haute dans l’histogramme indique donc qu’un grand nombre de pixels correspondent à l’altitude considérée.
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
im = Image.open('../Slides/figures/image.jpg')
channels = im.split()
z = np.array(channels[0])
N = z.size
n_classes = int(N**.5)
fig = plt.figure()
plt.clf()
fig.add_subplot(2, 1, 1)
plt.imshow(z, origin = "upper")
plt.colorbar()
fig.add_subplot(2, 1, 2)
plt.title('Histogramme')
plt.ylabel('Nombre de pixels')
plt.xlabel('Valeurs des pixels')
plt.hist(z.flatten(), bins=n_classes, histtype = "stepfilled")
plt.grid()
plt.show()
(Source code, png, hires.png, pdf)
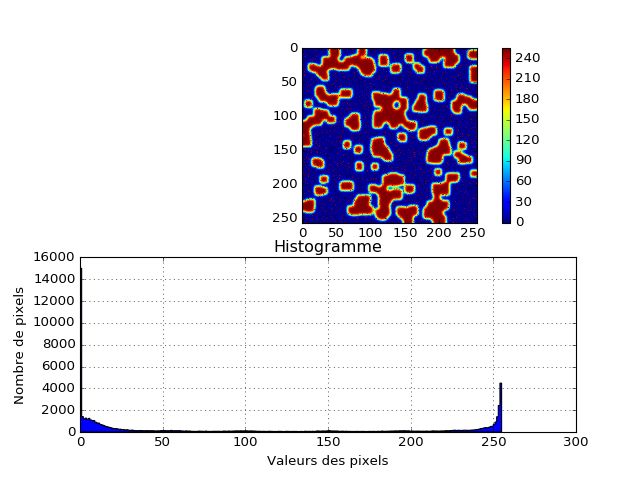
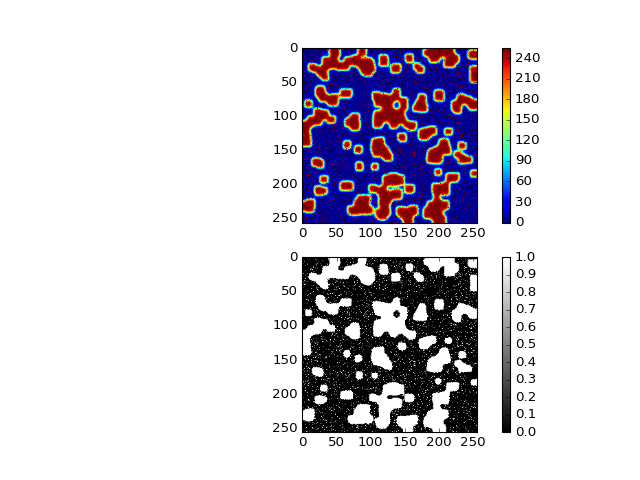
Seuillage¶
L’histogramme montre deux pics (\(Z = 20\) et \(Z = 230\)) correspondant à deux populations de pixels. Le seuillage consiste à transformer une image monochrome en une image binaire en appliquant un test booléen à chaque pixel. Une image binaire, c’est-à-dire formée de 0 et de 1 ou de Vrai et Faux est ainsi créé. Dans le cas présent, on peut alors chercher séparer les deux populations en effectuant un seuillage \(Z > 120\) :
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
from matplotlib import cm
im = Image.open('../Slides/figures/image.jpg')
channels = im.split()
z = np.array(channels[0])
seuil = 150.
zs = z > seuil
fig = plt.figure()
plt.clf()
fig.add_subplot(2, 1, 1)
plt.imshow(z, origin = "upper")
plt.colorbar()
fig.add_subplot(2, 1, 2)
plt.imshow(zs, origin = "upper", cmap = cm.gray)
plt.colorbar()
plt.show()
(Source code, png, hires.png, pdf)
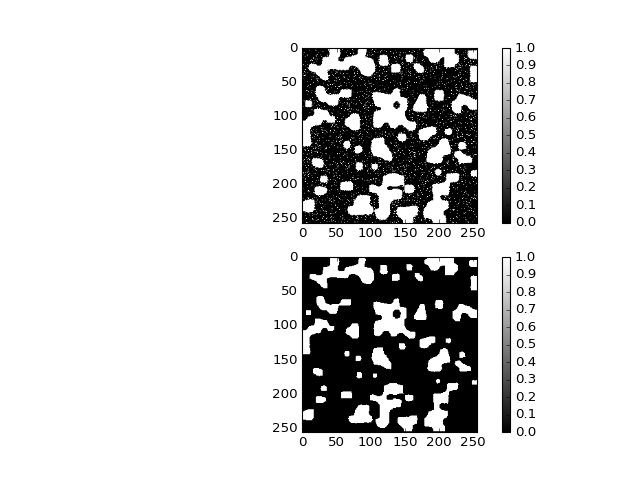
Erosion / Dilatation¶
On souhaite mesurer éliminer le bruit révélé par le seuillage effectué précédement. Pour ce faire, les outils issus de la morphologie mathématique tels que l’érosion et la dilatation sont particulièrement adaptés:
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
from matplotlib import cm
from scipy import ndimage
im = Image.open('../Slides/figures/image.jpg')
channels = im.split()
z = np.array(channels[0])
seuil = 150.
zs = z > seuil
zss = ndimage.morphology.binary_erosion(zs, structure=np.ones((3,3)))
fig = plt.figure()
plt.clf()
fig.add_subplot(2, 1, 1)
plt.imshow(zs, origin = "upper", cmap = cm.gray)
plt.colorbar()
fig.add_subplot(2, 1, 2)
plt.imshow(zss, origin = "upper", cmap = cm.gray)
plt.colorbar()
plt.show()
(Source code, png, hires.png, pdf)
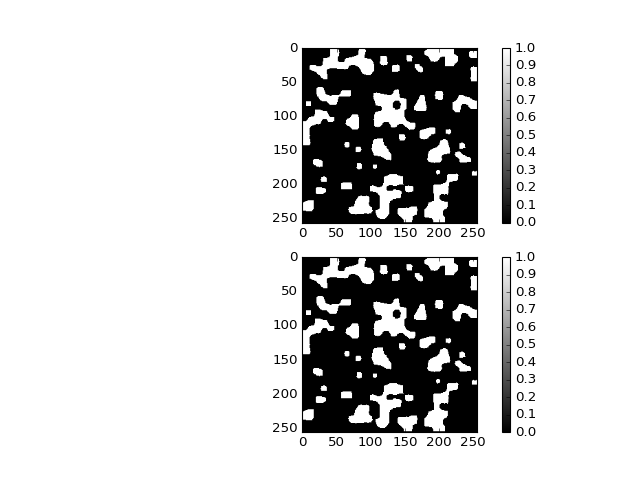
Pour restaurer la surface des zones partiellement érodées, on applique une dilatation:
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
from matplotlib import cm
from scipy import ndimage
im = Image.open('../Slides/figures/image.jpg')
channels = im.split()
z = np.array(channels[0])
seuil = 150.
zs = z > seuil
zse = ndimage.morphology.binary_erosion(zs, structure=np.ones((3,3)))
zsd = ndimage.morphology.binary_dilation(zse, structure=np.ones((3,3)))
fig = plt.figure()
plt.clf()
fig.add_subplot(2, 1, 1)
plt.imshow(zse, origin = "upper", cmap = cm.gray)
plt.colorbar()
fig.add_subplot(2, 1, 2)
plt.imshow(zse, origin = "upper", cmap = cm.gray)
plt.colorbar()
plt.show()
(Source code, png, hires.png, pdf)
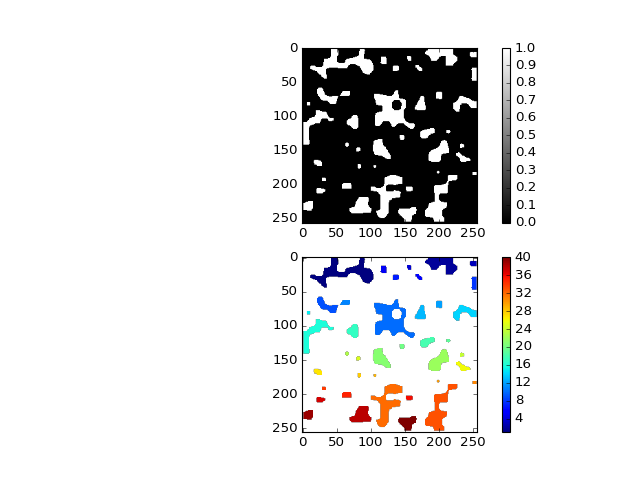
Comptage¶
Si on cherche maintenant a identifier individuellement les zones blanches mises en évidence lors du seuillage, il faut trouver tous les pixels appartenant à la terre \(Z = 1\) qui sont voisins. Le comptage de zones dans une image binaire peut se faire par des algorithmes dédiés . Voici un exemple:
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
from matplotlib import cm
from scipy import ndimage
im = Image.open('../Slides/figures/image.jpg')
channels = im.split()
z = np.array(channels[0])
seuil = 150.
zs = z > seuil
zse = ndimage.morphology.binary_erosion(zs, structure=np.ones((3,3)))
zsd = ndimage.morphology.binary_erosion(zse, structure=np.ones((3,3)))
zl, nombre = ndimage.measurements.label(zsd) # On compte les zones
zl = np.where(zl == 0, np.nan, zl)
fig = plt.figure()
plt.clf()
fig.add_subplot(2, 1, 1)
plt.imshow(zsd, origin = "upper", cmap = cm.gray)
plt.colorbar()
fig.add_subplot(2, 1, 2)
plt.imshow(zl, origin = "upper", cmap = cm.jet)
plt.colorbar()
plt.show()
(Source code, png, hires.png, pdf)
Recherche de contours¶
Si on cherche maintenant à trouver les contours des zones blanches. On peut combiner les opérateurs de dérivation:
from PIL import Image
import numpy as np
from matplotlib import pyplot as plt
from matplotlib import cm
from scipy import ndimage
im = Image.open('../Slides/figures/image.jpg')
channels = im.split()
z = np.array(channels[0])
seuil = 150.
zs = z > seuil
zse = ndimage.morphology.binary_erosion(zs, structure=np.ones((3,3)))
zsd = np.float64(ndimage.morphology.binary_erosion(zse, structure=np.ones((3,3))))
zgx = ndimage.sobel(zsd, axis=0, mode='constant')
zgy = ndimage.sobel(zsd, axis=1, mode='constant')
zsob = np.hypot(zgx, zgy)
fig = plt.figure()
plt.clf()
fig.add_subplot(2, 1, 1)
plt.imshow(zsd, origin = "upper", cmap = cm.gray)
plt.colorbar()
fig.add_subplot(2, 1, 2)
plt.imshow(zsob, origin = "upper", cmap = cm.gray)
plt.colorbar()
plt.show()
(Source code, png, hires.png, pdf)
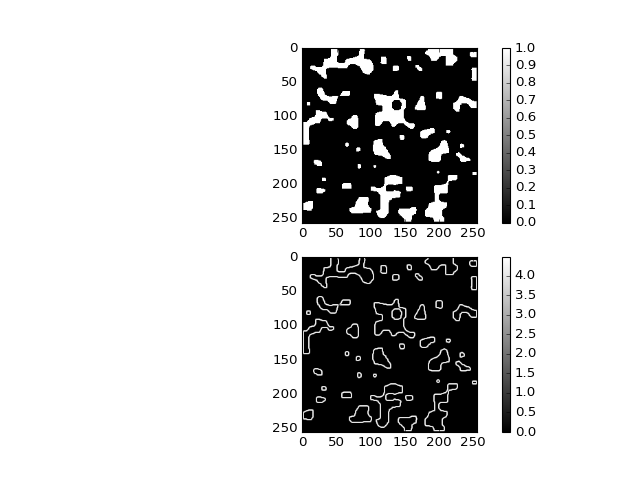
Les performances de la détection sont meilleures avec un filtre dédié comme le filtre de Canny .
Travaux Dirigés¶
On vous propose de travailler sur l’image suivante: (source ). On vous demande de faire un programme qui effectue les tâches suivantes:
- Lire l’image et la convertire au format numpy.array. Bien que d’aspect grisatre, l’image est en couleur et comporte donc 3 canaux. Il convient donc de choisir le canal le plus avantageux.
- Rogner séparer l’image en deux parties pour séparer le bandeau inférieur de l’image elle même.
- Tracer l’histogramme de l’image et en déduire un moyen de séparer les deux phases.
- Calculer la proportion de particules dans l’image.
- Compter les particules.
- Déterminer la taille moyenne des particules.